MatlabとPythonの連携その2
MatlabでPythonのライブラリを使えないかということで試してみます。
少し試しただけですがMatlab、Pythonともにインタプリタ言語なので割と親和性が高いように感じました。
MathWorks社のページ見ると2種類の方法があって
1.インラインコマンド・・・pyrun
2.Matlab上でPythonスクリプトファイルの実行・・・pyrunfile
1の方法はpyrun(code)をMatlab上で実行します。Pythonステートメントを一つ実行するたびにpyrunでくくる必要があるために、Pythonコードの規模が大きく複数行にまたがると結構めんどうかなという印象です。1行ずつ実行するにしても例えばimpotファイルなどは一度実行すればその後も有効っぽいので単純にpyrunが余分につくだけです。
2の方法はそもそもPythonで開発するので、そこで動くコードをスクリプトファイルとして保存しておけばpyrunfile(file)を実行しすればOKなので、コードを修正する場合はPythonで行えるので修正/確認が楽です。
ただ、だったら全部Pythonで書いても良いのかも知れませんSimulinkやほかのツールとの兼ね合いでMatlab使った方が楽な場合はMatlabを使えばいいと思います。
両方ともに言えるのはPythonが裏側で稼働しているためPython、ライブラリがインストールされてないと使えない、また変数(引数、戻り値)についてはお互いに引き渡す処理が必要です。特に難しいことではありませんが・・・
ちなみに下記が参考プログラムです。ニューラルネットワークでなくサポートベクターマシン(svm)を使ってアイリスの分類してみました。コードが数行で書けるので楽ちんです。またPythonはcコードを扱うよりもかなり楽です。
昔は振動や制御をFORTRAN、C、Matlabで力学的な解析してましたが、機械学習やディープラーニングについては全く素人ですがこうしたライブラリが提供されていると内容を深く理解せず、また複雑なコード書かずに実現できるので大変便利ですね。
なお、pyrun使った1の方法はMatlabとPythonを行ったり来たりする用途以外にはあまり使える場所がイメージできなかったので省略します。
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn import svm
import random
iris = datasets.load_iris()
iris_data = iris.data
iris_correct = iris.target
sw_data = iris_data[:100, :2]
iris_correct=iris_correct[:100]
clf = svm.SVC()
clf.fit(sw_data,iris_correct)
aa=clf.predict(sw_data)
ap = [[], []]
bp = [[], []]
for data in range(0,100):
if aa[data] < 0.5:
ap[0].append(data)
ap[1].append(data)
else:
bp[0].append(data)
bp[1].append(data)
print(sw_data[ap[1][:5]])
plt.scatter(sw_data[ap[0],0],sw_data[ap[0],1])
plt.scatter(sw_data[bp[0],0],sw_data[bp[0],1])
plt.show()
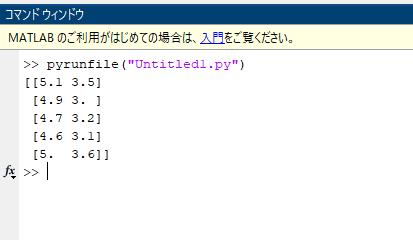
2の方法でPythonで動作確認済みのUntitled1.pyファイルをMatlab上でpyrunfile(“Untitled1.py”)と実行するとprint分はコマンドウインドウ上に、グラフはPythonのmatplotlibのウインドウに表示されました。
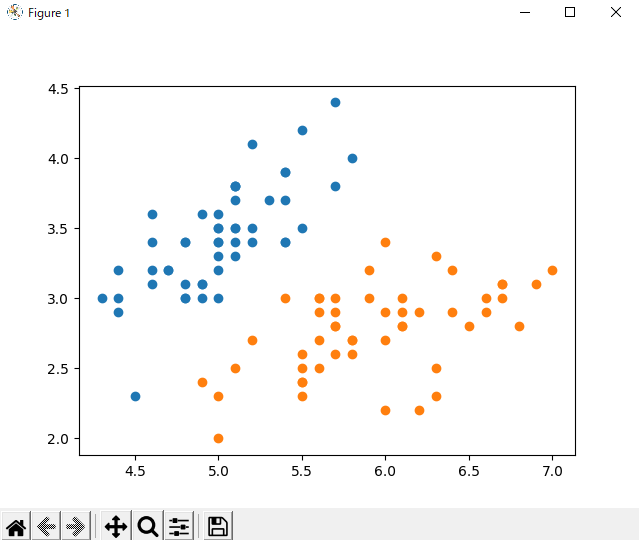
長くなりましたが思ったより簡単にMatlabのオプションツール使わずPythonとMatlabの組み合わせで結構機械学習使えそうです。